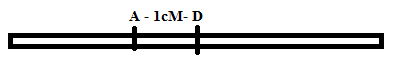|
|
Material destinado ao ensino e
aprendizagem de genética básica Difusão: laboratórios de biometria e bioinformática da Universidade Federal de Viçosa Site: www.ufv.br/dbg/biodata.htm Rede social: https://www.facebook.com/GBOLnews |
|
|
|
Para resolver os exercícios
recomendamos leituras adicionais nos sites: http://www.ufv.br/dbg/LabGen/gbol20.htm http://www.ufv.br/dbg/LabGen/gbol21.htm http://www.ufv.br/dbg/LabGen/GBOL20a.htm |
|
Ligação Fatorial
1)
Considere dois genes (A/a
e B/b) ligados, a uma distância de 20 unidades, e um gene C/c, independente. Em
relação a um triplo-heterozigoto em repulsão, responda:
a)
Quantos e quais os
diferentes gametas produzidos pelo indivíduo?
Como o indivíduo é um heterozigoto em repulsão (maior
probabilidade de um gene dominante e um recessivo se juntarem no gameta), então
ele é descrito da seguinte forma Ab//aB
Cc:
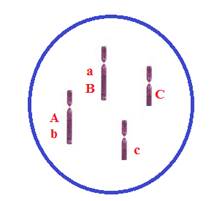
Assim, são formados:
|
Combinação |
Gameta |
Freqüência |
|
Ab – Parental C c |
AbC Abc |
P (1/2) P (1/2) |
|
aB – Parental C c |
aBC aBc |
P (1/2) P (1/2) |
|
AB – Recombinante C c |
ABC Abc |
R (1/2) R (1/2) |
|
ab – Recombinante C c |
abC abc |
R (1/2) R (1/2) |
Sendo,
P = (1-d)/2
R = d/2
2P + 2R = 1
d= distância
entre os dois genes (expressa em probabilidade)
Portanto teremos oito gametas diferentes.
b) Qual a
probabilidade de o indivíduo produzir gametas com a seguinte constituição
genotípica: AbC, AB e aC?
P(Ab C) = P(Ab) P(C)
= P (1/2) = [(1-d)/2] (1/2) = [(1-0,2)/2](1/2) = (0,4)(1/2) = 0,2
P(AB) = R = d/2 = 0.2/2 = 0,10
P(aC) = P(a) P(C) = (1/2) (1/2) = ¼ = 0,25
2)
Em tomate, dois genes
atuam da seguinte maneira: D/d - plantas anãs/altas; B/b - caule
liso/pubescente. Da autofecundação de um F1 (duplo-heterozigoto) surgiu a
descendência: anã, lisa - 5100; anã, pubescente - 2432; alta, lisa - 2422;
alta, pubescente - 46. Teste, pelo qui-quadrado, a hipótese de que os genes são
independentes e defina:
3)

a) valor do
qui-quadrado;
Para
encontrar o valor do qui-quadrado, teremos que relacionar uma tabela com a
quantidade de indivíduos que foi observada e a que está sendo esperada de
acordo com a proporção fenotípica.
Ho.: Genes são independentes
Cruzamento:
F1 (AaBb) autofecundado
|
Fenótipo |
Classe Fenotípica |
Observado |
Esperado (Sob Ho) |
(O-E)2/E |
|
Anã, lisa |
D-B- |
5100 |
(9/16)10000=5625 |
49 |
|
Anã, pubescente |
D-bb |
2432 |
(3/16)10000=1875 |
165,47 |
|
Alta, lisa |
ddB- |
2422 |
(3/16)10000=1875 |
159,58 |
|
Alta, pubescente |
ddbb |
46 |
(1/16)10000=625 |
536,39 |
|
|
|
10.000 |
|
X2c= 910,43 |
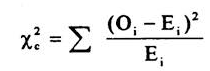
Portanto
o qui-quadrado é de 910,43.
b) grau de liberdade e
nível de significância estimado;
Como
temos 4 fenótipos, os graus de liberdade serão: Gl = n – 1 => 4-1= 3 Graus
de liberdade
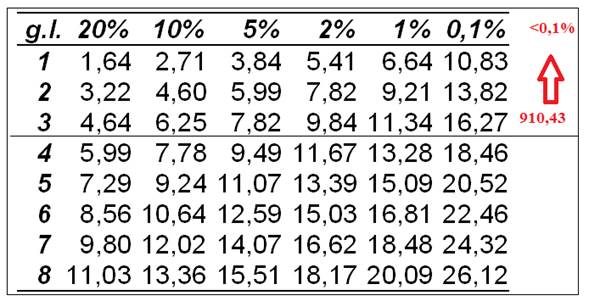
O nível
de significância será: a <0.001
c) Defina nível de
significância;
Nível de
significância estimado é o máxima probabilidade de erro, ao rejeitar Ho.
d) admitindo que os
genes D/d e B/b estão ligados, qual o genótipo da F1?
Para
resolver esta questão o leitor deverá ter o conhecimento sobre a frequência
esperada do homozigoto recessivo obtido da autofecundação de um
duplo-heterozigoto. Assim é esperado
|
Casos |
Genótipo do F1 |
Freq. esperada |
Estimador da
Freq. Esperada do homozigoto recessivo |
|
Genes
independentes |
Dd Bb |
1/16 = 0,0625 |
P(ddbb) = P(db) P(db) = (1/4)(1/4) = 1/6 |
|
Genes ligados –
F1 em aproximação |
DB // db |
Maior que 1/16 |
P(ddbb) = P(db) P(db) = P.P = [(1-d)/2][(1-d)/2} = (1-d)2/4 |
|
Genes ligados –
F1 em repulsão |
Db // dB |
Menor que 1/16 |
P(ddbb) = P(db) P(db) = R.R = (d/2) (d/2) = d2/4 |
No
exemplo considerado, temos:
F(dd bb)= 46\10.000= 0,0046
Como 0,0046 < 0,0625, consequentemente os genótipos da F1 estão em repulsão. Por isso o genótipo de F1 é Db//dB.
e) qual a distância
entre os genes?
Usando o
estimador apropriado, que neste caso é aquele aplicável à situação do F1 duplo-heterozito
estar em repulsão, temos:
P(ddbb) = P(db) P(db) =
R.R = (d/2) (d/2) = d2/4
No
exemplo
P(ddbb) = P(db) P(db) = R.R = (d/2) (d/2) = d2/4 = 46/10000
Assim,
![]() = 0,1356 ou 13,56% = 13,56cM
= 0,1356 ou 13,56% = 13,56cM
f) qual a
porcentagem de quiasmas entre estes genes que seria observada em exames
citológicos de indivíduos desta espécie?
Sabemos
que:
% quiasmas= 2x (%Recombinantes)
%
quiasmas= 2x (13,56) = 27,12%
3) Considere três genes: A/a, B/b e C/c. Os
genes A/a e B/b estão ligados com distância igual a 16 unidades. O gene C/c é
independente. Dado um indivíduo X triplo-heterozigoto, em aproximação para os
genes ligados, responda:
a)
Qual a frequência de gametas ABC produzidos por X?
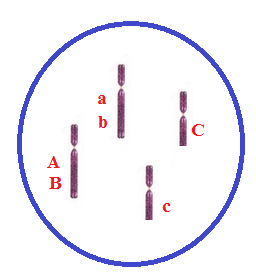
Faremos
a frequência dos gametas ABC:
P(ABC) = P(AB) x P(C) =
P (1/2) = [(1-d)/2](1/2) =[(1-0.16)/2](1/2)= -,21 = 21%
b) Qual a
frequência de indivíduos aabb obtidos a partir da autofecundação de X?
Para
essa questão devemos apenas multiplicar a frequência dos gametas ab no mesmo
indivíduo. Como a distância é 16 unidades, temos a seguinte frequência:
P(aabb) = P(ab) x P(ab) = PxP
= [(1-d)\2]x[(1-d)\2]= [(1-0,16)\2]x[(1-0,16)\2]= 0,1764= 17,64%
c) Qual a
frequência de indivíduos aacc obtidos a partir da autofecundação de X?
Esse
caso é bem interessante visto que o gene a e o gene c estão separados nos
cromossomos. Portanto a frequência de cada um é 0,5. Com isso temos:
P(aacc) = P(ac) x P(ac) = (1/4)(1/4) = 1/16 = 0,0625=
6,25%
d) Quais seriam as
respostas destas mesmas questões (a,b e c) se o indivíduo X fosse
triplo-heterozigoto em repulsão para os genes ligados?
1 ->
P(ABC) = P(AB) x P(C) = R (1/2) = (d\2)x (1/2)= (0,16/2) x (1/2)=0,04= 4%
2 -> P(aabb) = P(ab) x P(ab) = RxR = (d/2) x (d/2)
= (0,16/2) x (0,16/2) = 0,0064= 0,64%
3 -> P(aacc) = P(ac) x P(ac) = (1/4)(1/4) = 1/16 =
0,0625= 6,25%
4) Em uma
espécie vegetal, três genes atuam da seguinte maneira: A/a - planta alta/anã;
B/b - sementes lisas/rugosas; C/c - flores roxas/brancas. Quando plantas F1
triplo-heterozigotas foram submetidas ao cruzamento-teste, surgiu à
descendência: 302 altas, rugosas, roxas; 91 altas, lisas, roxas; 92 anãs,
rugosas, roxas; 17 anãs, lisas, roxas; 18 altas, rugosas, brancas; 94 altas,
lisas, brancas; 93 anãs, rugosas, brancas; e 293 anãs, lisas, brancas.
a) Qual a ordem dos
genes?
O cruzamento foi realizado por triplo-heterozigotos com plantas
recessivas (cruzamento-teste), ou seja:
Aa Bb Cc X aa bb cc
Esse cruzamento resultou na seguinte tabela dada pelo exercício:
|
Altura |
Semente |
Cor |
Observado |
Tipo
de classe* |
|
altas |
rugosas |
roxas |
302 |
P |
|
altas |
lisas |
roxas |
91 |
|
|
anãs |
rugosas |
roxas |
92 |
|
|
anãs |
lisas |
roxas |
17 |
Rd |
|
altas |
rugosas |
brancas |
18 |
Rd |
|
altas |
lisas |
brancas |
94 |
|
|
anãs |
rugosas |
brancas |
93 |
|
|
anãs |
lisas |
brancas |
293 |
P |
|
Total |
|
|
1000 |
|
A segregação do tipo 2P:2R1:2R2:2Rd é típica de três-genes ligados,
assim considera-se:
*P : classe fenotípica formada a partir do gameta paternal do
triplo-heterozigoto. É a classe mais frequente.
Rd: classe fenotípica formada a partir do gameta que tem cromática
com dupl-recombinação. É a classe menos frequente.
Para se estabelecer a ordem entre os genes compara-se uma classe P
com outra Rd e identifica-se o gene que ocupa a posição intemediária.
|
Paternais |
Duplo
Recombinantes |
Comparação |
Conclusão: O gene
que ocupa a posição intermediária é: |
|
altas rugosas roxas |
anãs lisas
roxas |
≠≠= |
C/c |
|
altas rugosas roxas |
altas rugosas brancas |
==≠ |
C/c |
|
anãs lisas brancas |
anãs lisas
roxas |
== ≠ |
C/c |
|
anãs lisas brancas |
altas rugosas brancas |
≠≠= |
C/c |
Vendo que
há uma recombinação entre os parentais, e que comparando eles com os duplo
recombinantes poderemos encontrar o gene central. Assim, o gene C\c ocupa a posição
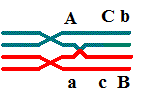
intermediária, e a ordem correta será:
A\a-C\c-
B\b ou B\b- C\c- A\a.
Com esta informação devemos acrescentar uma coluna à tabela de
dados referente ao gameta do triplo-heterozigoto que originou cada classe
fenotípica, na ordem correta. Ou seja:
|
Altura |
Semente |
Cor |
Observado |
Tipo
de classe* |
Gameta
do F1 em ordem correta |
|
altas |
rugosas |
roxas |
302 |
P |
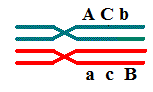
A C b |
|
altas |
lisas |
roxas |
91 |
|
A C B |
|
anãs |
rugosas |
roxas |
92 |
|
a C b |
|
anãs |
lisas |
roxas |
17 |
Rd |
a C B |
|
altas |
rugosas |
brancas |
18 |
Rd |
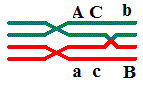
A c b |
|
altas |
lisas |
brancas |
94 |
|
A c B |
|
anãs |
rugosas |
brancas |
93 |
|
a c b |
|
anãs |
lisas |
brancas |
293 |
P |
a c B |
|
Total |
|
|
1000 |
|
|
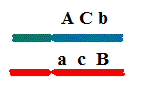
b) Qual o genótipo do F1?
O
genótipo do F1 é reconstituído por meio das informações dos gametas paternais
(na ordem correta), identificados pela sua
maior frequência. Assim, conclui-se que F1, triplo-heterozigoto,
apresenta genótipo:
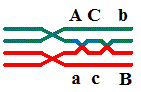
ACb\\acB.
c) Qual a distância entre os genes?
Inicialmente
precisamos definir quais seriam os gametas do triplo-heterozigoto que contém
cromátides resultantes de permita na região 1 (R1), entre os genes A/a e C/c e
na região 2, entre os genes C/c e B/b.
Assim, temos:
|
Altura |
Semente |
Cor |
Observado |
Tipo
de classe* |
Gameta
do F1 em ordem correta |
Origem |
|
altas |
rugosas |
roxas |
302 |
P |
A C b |
|
|
altas |
lisas |
roxas |
91 |
R2 |
A C B |
|
|
anãs |
rugosas |
roxas |
92 |
R1 |
a C b |
|
|
anãs |
lisas |
roxas |
17 |
Rd |
a C B |
|
|
altas |
rugosas |
brancas |
18 |
Rd |
A c b |
|
|
altas |
lisas |
brancas |
94 |
R1 |
A c B |
|
|
anãs |
rugosas |
brancas |
93 |
R2 |
a c b |
|
|
anãs |
lisas |
brancas |
293 |
P |
a c B |
|
|
Total |
|
|
1000 |
|
|
|
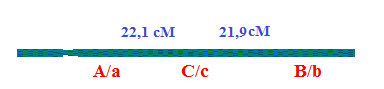
Com isso temos:
d(A\a-C\c) = d1 = (R1 + R1 +Rd
Rd)/Total = (92 + 94 + 17 + 18)\ 1000 = 0,221 ou 22,1cM
d(C\c- B\b) = d2 = (R2 + R2 +Rd
Rd)/Total = (91 + 93 + 17 + 18)\
1000 = 0,219 ou 21,9 cM
d) Qual o coeficiente de interferência para a região cromossômica
estudada?
A interferência e coincidência são estimadas a partir de
informações sobre o crossing-over duplo observado (CODO) e esperado sob
hipótese de interferência nula (CODE).
CODE = (d1/100) x(d2/100)x 100 = (22,1 x 21,9)\100=
4,84%
CODO= [(Rd+Rd)x100]/Total =
[(17+18)x100]\ 10000= 3,5%
Portanto,
Co= CODO/CODE = 3,5\4,84=0,723
Ocoeficiente de interferência
é:
I= 1 – Co= 1-0,723=0,2768
5) Em Drosophila, três genes atuam da seguinte
maneira: B/b: corpo de coloração selvagem/preto; V/v: asas
selvagens/vestigiais; e M/m: olhos de coloração selvagem/marrom. Quando moscas
F1 triplo-heterozigotas foram submetidas ao cruzamento-teste, surgiu a
descendência:
|
Corpo |
|
|
|
Asas |
|
|
|
Olhos |
|
|
a) Qual a ordem dos genes?
O cruzamento foi realizado por triplo-heterozigotos com plantas
recessivas (cruzamento-teste), ou seja:
Bb Vv Mm x bb vv mm
Esse cruzamento resultou na seguinte tabela dada pelo exercício:
|
Corpo |
Asaa |
Olhos |
Observado |
Tipo de classe* |
|
Selvagem |
Selvagem |
Selvagem |
354 |
P |
|
Preto |
Vestigial |
Marrom |
362 |
P |
|
Preto |
Selvagem |
Marrom |
72 |
|
|
Preto |
Vestigial |
Selvagem |
5 |
Rd |
|
Preto |
Selvagem |
Selvagem |
70 |
|
|
Selvagem |
Vestigial |
Selvagem |
68 |
|
|
Selvagem |
Vestigial |
Marrom |
61 |
|
|
Selvagem |
Selvagem |
Marrom |
8 |
Rd |
|
Total |
|
|
1000 |
|
A segregação do tipo 2P:2R1:2R2:2Rd é típica de três-genes ligados, assim
considera-se:
*P : classe fenotípica formada a partir do gameta paternal do
triplo-heterozigoto. É a classe mais frequente.
Rd: classe fenotípica formada a partir do gameta que tem cromática
com dupl-recombinação. É a classe menos frequente.
Para se estabelecer a ordem entre os genes compara-se uma classe P
com outra Rd e identifica-se o gene que ocupa a posição intermediária.
|
Paternais |
Duplo
Recombinantes |
Comparação |
Conclusão: O gene
que ocupa a posição intermediária é: |
|
Selvagem Selvagem Selvagem |
Preto Vestigial Selvagem |
= = ≠ |
M\m |
|
Selvagem Selvagem Selvagem |
Selvagem Selvagem Marrom |
= = ≠ |
M\m |
|
Preto Vestigial Marrom |
Preto Vestigial Selvagem |
= = ≠ |
M\m |
|
Preto Vestigial Marrom |
Selvagem Selvagem Marrom |
≠ ≠ = |
M\m |
Vendo
que há uma recombinação entre os parentais, e que comparando eles com os duplo
recombinantes poderemos encontrar o gene central. Assim, o gene M\m ocupa a posição
intermediária, e a ordem correta será:
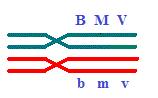
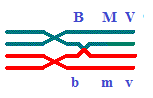
B\b-M\m-
V\v ou V\v- M\m- B\b.
Com esta informação devemos acrescentar uma coluna à tabela de
dados referente ao gameta do triplo-heterozigoto que originou cada classe
fenotípica, na ordem correta. Ou seja:
|
Corpo |
Asa |
Olhos |
Observado |
Tipo
de classe* |
Gameta
do F1 em ordem correta |
|
Selvagem |
Selvagem |
Selvagem |
354 |
P |
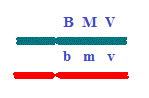
B M V |
|
Preto |
Vestigial |
Marrom |
362 |
P |
b m v |
|
Preto |
Selvagem |
Marrom |
72 |
|
b m V |
|
Preto |
Vestigial |
Selvagem |
5 |
Rd |
b M v |
|
Preto |
Selvagem |
Selvagem |
70 |
|
b M V |
|
Selvagem |
Vestigial |
Selvagem |
68 |
|
B M v |
|
Selvagem |
Vestigial |
Marrom |
61 |
|
B m v |
|
Selvagem |
Selvagem |
Marrom |
8 |
Rd |
B m V |
|
Total |
|
|
1000 |
|
|
b) Qual o genótipo do F1?
O
genótipo do F1 é reconstituído por meio das informações dos gametas paternais
(na ordem correta), identificados pela sua
maior frequência. Assim, conclui-se que F1, triplo-heterozigoto, apresenta
genótipo:
B M V \\ b m v.
|
|
|
c) Qual a distância entre os genes?
Inicialmente precisamos
definir quais seriam os gametas do triplo-heterozigoto que contém cromátides
resultantes de permita na região 1 (R1), entre os genes B/b e M/m e na região
2, entre os genes M/m eV/v.
Assim, temos:
|
Altura |
Semente |
Cor |
Observado |
Tipo
de classe* |
Gameta
do F1 em ordem correta |
Origem |
|
Selvagem |
Selvagem |
Selvagem |
354 |
P |
B M V |
|
|
Preto |
Vestigial |
Marrom |
362 |
P |
b m v |
|
|
Preto |
Selvagem |
Marrom |
72 |
R2 |
b m V |
|
|
Preto |
Vestigial |
Selvagem |
5 |
Rd |
b M v |
|
|
Preto |
Selvagem |
Selvagem |
70 |
R1 |
b M V |
|
|
Selvagem |
Vestigial |
Selvagem |
68 |
R2 |
B M v |
|
|
Selvagem |
Vestigial |
Marrom |
61 |
R1 |
B m v |
|
|
Selvagem |
Selvagem |
Marrom |
8 |
Rd |
B m V |
|
|
Total |
|
|
1000 |
|
|
|
Com isso temos:
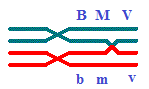
d(B/b – M/m) = d1 = (R1 + R1 +Rd
Rd)/Total = (70+ 61+ 8+5)\ 1000 = 0,144 ou 14,4cM
d(M/m – V/v) = d2 = (R2 + R2 +Rd
Rd)/Total = (72+ 68+ 8+5)\ 1000 = 0,153 ou 15,3cM

d) Qual o coeficiente de interferência para a
região estudada?
A interferência e coincidência são estimadas a partir de
informações sobre o crossing-over duplo observado (CODO) e esperado sob
hipótese de interferência nula (CODE).
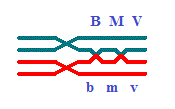
CODE = (d1/100) x(d2/100)x 100 = (14,4 x 15,3)\100=
2,2032%
CODO= [(Rd+Rd)x100]/Total =
[(8+5)x100]\ 10000= 1,3%
Portanto,
Co= CODO/CODE = 2,2032\1,3=0,59
O coeficiente de
interferência é:
I= 1 – Co= 1-0,59=
0,41
6) Considere três genes (A/a, B/b e C/c) ligados (distância entre
A/a e B/b igual a 10 unidades, distância entre B/b e C/c de 20 unidades e
interferência igual a 0,6). Os genes D/d e E/e também estão ligados com
distância igual a 16 unidades.
Dado um indivíduo cuja constituição genotípica é ilustrada na célula
a seguir, responda:
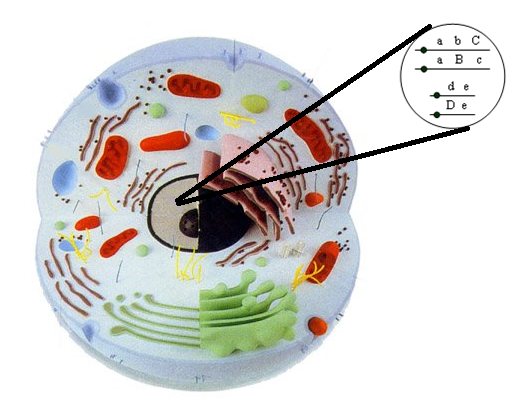
a) Qual a frequência de gametas aBD?
Em relação aos três genes ligados, podemos admitir que são
produzidos, por um triplo-heterozigoto, oito gametas com frequência P,P,
R1,R1,R2,R2,Rd e Rd. Devemos encontrar estes valores a partir das informações
sobre as distâncias e sobre a interferência, da seguinte maneira:
- Estimativa de RD – é estimado a partir dos
valores esperados de CODO
Sabemos que CODE = (d1/100)x(d2/100)x100 = (10/100)(20/100)100= 2%
Sabemos que Co = CODO/CODE e que I + Co = 1
Então
CODO = Co CODE = (1 – I) CODE = (1 -0,6) (2%) = 0,8%
Por fim, utilizamos a expressão:
CODO = (Rd + Rd)x100/Total
Para fins de predição admitimos Total = 100
Logo
CODO = 2Rd
Rd = CODO/2
Rd = 0,8/2 = 0,4%
- Estimativa de
R1 – é estimada a partir dos valores de d1
Sabemos que d1 =[ (R1 + R1 + Rd + Rd)x100]/Total
Então (se Total = 100)
d1 = 2R1 + 2Rd = 2R1 + CODO
logo
R1 = (d1 – CODO)/2
R1 = (10- 0,8)/2 = 4,6%
- Estimativa de R2 – é estimada a partir
dos valores de d2
Sabemos que d2 =[ (R2 + R2 + Rd + Rd)x100]/Total
Então (se Total = 100)
d2 = 2R2 + 2Rd = 2R2 + CODO
logo
R2 = (d2 – CODO)/2
R2 = (20- 0,8)/2 = 9,6%
- Estimativa de P– é estimada por diferença
Sabemos que:
2P + 2 Rd + 2R1 + 2R2=100%
2P + 0,8 + 2(4,6)+
2(9,6)=100%
2P+ 29,2=100%
P=35,4%
Podemos
agora encontrar a resposta:
P(aBD)
= P(aB) P(D)
O leitor
deverá saberá origem, em termos de ocorrência de permuta, de gametas contendo
cromátides aB. Os gametas de um indivíduo abC//aBc será:
|
Gametas |
abC |
aBc |
abC |
aBc |
abc |
aBC |
aBC |
abc |
|
Frequencia |
P |
P |
R1 |
R1 |
R2 |
R2 |
Rd |
Rd |
Conclui-se
que:
P(aB)=P+ Rd +R1 + R2
F(aB)=35,4+ 0,4+9,6+4,6
F(aB)=50%
Como
o gameta D tem 0,5 de probabilidade de ser formado, temos a seguinte
multiplicação de probabilidade:
P(aBD)=P(aB)x
P(D)= 0,5x0,5=0,25
(*) Este problema poderia ser resolvido ignorando o gene C/c
(não mencionado na questão) e admitindo
que apenas dois tipos de gametas são formados: aB e ab com frequência de 50%
cada um dele, dispensando todos os cálculos mencionados anteriormente.
Entretanto, como veremos no exemplo a seguir esta forma apresentada é de de
aplicação geral.
b) Qual a frequência de gametas BCD?
P(BCD)=P(BC)x
P(D)
Para o cálculo da probabilidade de gametas BC
consideramos as possibilidades:
|
Gametas |
abC |
aBc |
abC |
aBc |
abc |
aBC |
aBC |
abc |
|
Frequencia |
P |
P |
R1 |
R1 |
R2 |
R2 |
Rd |
Rd |
P(BC) = R2 + Rd = 9,6% + 0,4% = 10%
Logo,
P(BCD)=P(BC)x
P(D) = (10%)(1/2) = 5%
c) abcde?
P(abcde) =
P(abc)P(de)
Para os genes A/a, B/b e C/c temos
|
Gametas |
abC |
aBc |
abC |
aBc |
abc |
aBC |
aBC |
abc |
|
Frequencia |
P |
P |
R1 |
R1 |
R2 |
R2 |
Rd |
Rd |
P(abc) = R2 + Rd 9,6% + 0,4% = 10%
Para os genes D/d e E/e
Apenas dois gametas são formados a partir de Ddee que são ½ De e ½
de
P(abcde)=P(abc)x P(de)=
(10%) x 0,5=5%
7) A distância
entre dois genes (A/a e B/b) é de 30 unidades. Considerando o cruzamento entre
duplo-heterozigotos, envolvendo um indivíduo X em aproximação e um indivíduo Y
em repulsão, responda:
|
X:
|
|
Y:
|
a) Qual a probabilidade de o indivíduo X
produzir gametas aB?
O indivíduo X está em aproximação, ou seja, o arranjo nos cromossomos está na seguinte forma AB//ab. Portanto, o gametas aB, cuja distância dos genes sendo de 30 unidades, é recombinante:
P(aB) = R = d/2=0,3/2=0,15
b) Qual a probabilidade de o indivíduo Y
produzir gametas Ab?
O indivíduo Y possuí o arranjo em repulsão, ou seja ele é Ab//aB. O gameta Ab é do teipo paternal, ou seja::
P(Ab)= P = (1-d)/2 = (1-0,3)/2=0,7/2=0,35
c) Qual a probabilidade de surgirem descendentes
homozigotos recessivos (ab//ab) a partir deste cruzamento?
P(ab//ab)= P(ab de X) x P(ab de Y) = P x R = [(1-d)/2] x (d/2) = [(1-0,3)/2)] x (0,3/2)
= 0,0525 ou 5,25%
8) Em
tomate, o fruto é redondo (O-) ou alongado (oo), e a inflorescência, simples
(S-) ou composta (ss). Uma planta F1, duplo-heterozigota, foi submetida ao
cruzamento-teste, proporcionando os seguintes descendentes: 38 alongados
simples, 170 redondos simples, 166 alongados compostos e 46 redondos compostos.

a)
Os genes O/o e S/s
estão ligados? (justifique a sua resposta);
Inicialmente vamos formular a hipótese de que os genes são
independentes
Ho: Se os genes O\o e S\s são independentes com
segregação 1:1:1:1
Cruzamento: F1:
Oo Ss x Testador:
oo ss
Progênie:
|
Fenótipo |
Gameta do F1 |
Observado |
Esperado sob Ho |
(O – E)2/E |
|
Alongado,simples |
o S |
38 |
( ¼ )420 = 105 |
42,75 |
|
Redondo,simples |
O S |
170 |
( ¼ )420 = 105 |
40,24 |
|
Alongado,composta |
o s |
166 |
( ¼ )420 = 105 |
35,44 |
|
Redondo,composta |
O s |
46 |
( ¼ )420 = 105 |
33,15 |
|
Total |
|
420 |
|
151,58 |
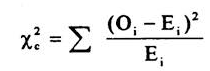
Portanto
o qui-quadrado é de 151,58
Como
temos 4 fenótipos, os graus de liberdade serão: Gl = n – 1 => 4-1= 3 Graus
de liberdade
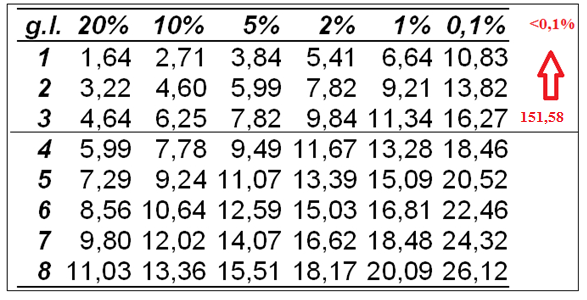
Conclusão:
Como a <
0,001 é pequeno decidimos pela rejeição de Ho. Ou seja há evidencias que os
genes O/o e S/s estão ligados.
b) Qual o
genótipo do F1?
Os gametas da F1 são estabelecidos a partir de
seus gametas do tipo paternal.
|
Fenótipo |
Gameta do F1 |
Observado |
Classificação dos
gametas |
|
Alongado,simples |
o S |
38 |
R |
|
Redondo,simples |
O S |
170 |
P |
|
Alongado,composta |
o s |
166 |
P |
|
Redondo,composta |
O s |
46 |
R |
|
Total |
|
420 |
|
(*) P: gameta paternal de maior frequência
R: gameta recombinante de menor frequencia
Como as classes paternais, de maior frequência,
se referem aos gametas OS e os, conclui-se que o duplo-heterozigoto envolvido apresenta
os genes ligados em fase de aproximação, de forma que o genótipo é representado
por OS\\os.
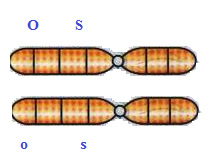
c) Se os
genes estiverem ligados, calcule a distância entre O/o e S/s.
O cruzamento teste é bastante útil em estudos de ligação fatorial pois as frequências dos descendentes refletem a frequência dos gametas produzidos pelo F1. Assim, podemos usar a definição de que a distância entre os genes é igual a frequência de gametas recombinantes entre os genes analisados, ou seja;
d= ∑(gametas R)/Total x
100 =
(46+38)/420 x100= 20% =20cM
9) Assinale verdadeiro (V) ou falso (F):
Para responder as questões abaixo lembre que:
1.
Dois genes ligados
estão em um mesmo cromossomossomos
2.
Dois genes
pertencentes a cromossomos diferentes são considerados independentes
3.
Dois genes segregam
independentemente quando P(AB) = P(A)P(B)
4.
Um
duplo-heterozigoto (AaBb) tem P(AB) = P(A).P(B) = 1/4
5.
Um
duplo-heterozigoto (AB//ab) tem P(AB) = P(A).P(B/A) = (1-d)/2 (será igual a ¼ apenas quando d = 50cM =
0,50)
6.
Um
duplo-heterozigoto (Ab//aB) tem P(AB) = P(A).P(B/A) = d/2 (será igual a ¼ apenas quando d = 50cM =
0,50)
a- genes independentes são aqueles localizados
em cromossomos distintos (V)
b- genes ligados são aqueles localizados em um
mesmo cromossomo (V)
c- genes independentes têm distribuição
independente (V)
d- genes ligados não têm distribuição
independente (F ) – Podem ter distribuição independente quando a distância eles for
igual ou superior a 50cM.
e- se dois genes têm distribuição independente,
eles são independentes (F) – Dois genes cuja
distância é igual ou superior a 50cM terá segregação independente apesar de
serem ligados.
f- se dois genes não têm distribuição independente,
eles são ligados (V )
g- dois genes completamente ligados não têm
distribuição independente (V )
h- para que dois genes ligados tenham
distribuição independente, basta ocorrer crossing-over entre eles (F) – É necessário que a
porcentagem de crossing-over atinja 50%.
i- dois genes que têm
distribuição independente estão localizados em cromossomos distintos da espécie
(F)
– Não necessariamente, eles podem ter mais de 50 cM com isso
consideram independentes também.
10) Os
seis genes a seguir são do milho, sendo quatro do cromossomo 2 e dois do
cromossomo 8. Responda:
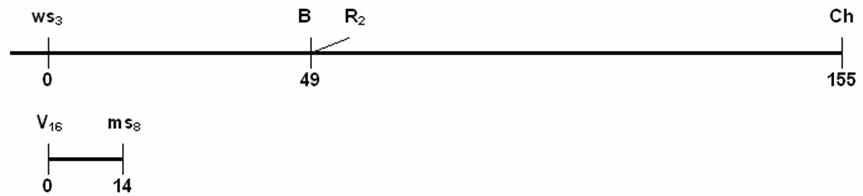
(a)
Indique dois completamente ligados.
Completamente ligados, ou seja, muito próximos. Resposta: os genes B e R2.
(b)
Indique dois incompletamente ligados.
São genes ligados e, portanto, pertencentes aos mesmo cromossomo: Resposta:
V16 e ms8
(c)
Indique dois independentes.
Genes pertencentes a cromossomos diferentes. Resposta: Ws3 e ms8 .
(d)
Indique dois ligados com distribuição independente.
São genes
que estão no mesmo cromossomo mas estão em uma distância maior que 50cM.
Resposta: Ws3 e Ch.
(e) Qual
deve ser o par de genes fisicamente mais próximos?
Genes B e R2
estão fisicamente muito próximos. Lembrar que as medidas apresentadas no
gráfico não medem a distância física mas a probabilidade de permuta entre os
genes.
(f) Qual
deve ser o par de genes fisicamente mais distantes?
Genes
Ws3 e Ch são os fisicamente mais distantes no mesmo cromossomo.
(g) Calcule
a proporção gamética dos seguintes duplo-heterozigotos:
B b Ms8 ms8
B R2 / b r2
Ws3 Ch / ws3 ch
V16
Ms8 / v16 ms8
Resposta:
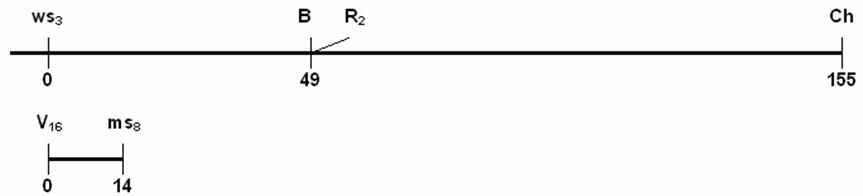
|
Duplo-heterozigoto |
B b Ms8 ms8 |
B R2 / b r2 |
Ws3 Ch / ws3 ch |
V16 Ms8 / v16 ms8 |
|
Distância |
Independentes |
0 cM |
Seg. Independ |
14cM |
|
Gametas: |
|
|
|
|
|
|
B Ms8 ¼ |
B R2 ½ |
Ws3 Ch ¼ |
V16 Ms8
P = (1-d)/2 = (1 – 0,14)/2 =
0,43 |
|
|
B ms8 1/4 |
b r2 ½ |
Ws3 ch ¼ |
v16 ms8
P = (1-d)/2 = (1 – 0,14)/2 =
0,43 |
|
|
b Ms8 ¼ |
|
ws3 Ch ¼ |
V16 ms8
R = d/2 = 0,14/2 = 0,07 |
|
|
b ms8 1/4 |
|
ws3 ch ¼ |
v16 Ms8
R = d/2 = 0,14/2 = 0,07 |
11) Em Drosophila melanogaster, os locos "yellow"
(Y/y) e "scute" (S/s) são completamente ligados. A distância entre os
locos "net" (N/n) e "plexus" (P/p) é de 100,5 unidades de
mapa. Considere uma fêmea de genótipo YySs e outra de genótipo NnPp. Indique a
frequência de gametas recombinantes que cada fêmea deve produzir.

Para a fêmea YySs em que os genes estão
completamente ligados, logo só haverá gametas paternais, com isto a porcentagem
de gametas recombinantes é de 0%.
Para a fêmea NnPp os seus genes estão em uma
distância de 100,5cM, logo a segregação ocorrerá de forma independente. Assim,
independente da fase de ligação (aproximação e repulsão) teremos:
NP
– ¼
Np
– ¼
nP
– ¼
np
– ¼
12) Em Drosophila melanogaster, a distância entre os
locos "stripe" (S/s) e "ebony" (E/e) é de 8,7 unidades de
mapa. Os genes recessivos s e e determinam
corpo com listras e corpo de cor ébano, respectivamente. Os genes dominantes S
e E condicionam corpo sem listras e corpo de cor cinza, respectivamente. Quais
as proporções genotípicas e fenotípicas esperadas na descendência do cruzamento
entre fêmeas duplo-heterozigotas em configuração "cys" (aproximação) com machos duplo-heterozigotos em
configuração "trans" (repulsão) ? (admita que não haja crossing-over
nos machos).

Ação
gênica:
|
|
Corpo |
Cor |
|
S – E – |
Sem
listra |
Cinza |
|
S – ee |
Sem
listra |
ébano |
|
ss E – |
Com
listra |
cinza |
|
ss ee |
Com
listra |
ébano |
Cruzamento:
|
|
x |
|
|
S E //
s e |
|
S e //
s E |
Descendência
|
Fêmea\macho |
Se (1/2) |
sE (1/2) |
|
SE(P) |
SS
Ee ( ½ P) |
Ss EE
( ½ P) |
|
Se(R) |
SS
ee ( ½ R) |
Ss Ee
( ½ R) |
|
sE(R) |
Ss
Ee ( ½ R) |
ss EE (
½ R)) |
|
se(P) |
Ss
ee ( ½ P) |
ss Ee
( ½ P) |
Proporções fenotípicas:
S-E- = 1/2P +
1/2P + 1/2R + 1/2R = P + R = (1-d)/2 +
d/2 = 0,5
S-ee = 1/2P + 1/2R = ½(P + R) = ½
(0,5) = 0,25
ssE- = 1/2P + 1/2R = ½(P + R) = ½
(0,5) = 0,25
ssee= 0
13) O
daltonismo "deutan" é um caráter determinado por gene recessivo
ligado ao sexo. A doença "retinitis pigmentosum" (cegueira completa
ou parcial) é determinada por gene dominante parcialmente ligado ao sexo. Uma
mulher não daltônica e com retinite, cuja mãe é daltônica e sem retinite e o
pai não daltônico e com retinite, homozigoto, casa-se com um homem daltônico e
sem retinite. Considerando que a distância entre os dois locos, no X, é de 10
unidades de mapa, responda:
(a) Qual
a proporção genotípica esperada na descendência?
Daltonismos
|
Mulheres |
|
|
Homens |
|
|
XD
XD |
Normal |
|
XD
Y |
Normal |
|
XD
Xd |
Normal |
|
Xd
Y |
Daltônico |
|
Xd
Xd |
Daltônica |
|
|
|
Retinite
|
Mulheres |
|
|
Homens |
|
|
XR
XR |
Retinite |
|
XR
YR |
Retinite |
|
XR
Xr |
Retinite |
|
XR
Yr |
Retinite |
|
Xr
Xr |
Normal |
|
Xr
YR |
Retinite |
|
|
|
|
Xr
Yr |
Normal |
Cruzamento:
Essa imagem irá ajudar a encontrar as proporções das futuras gerações em relação ao daltonismo.
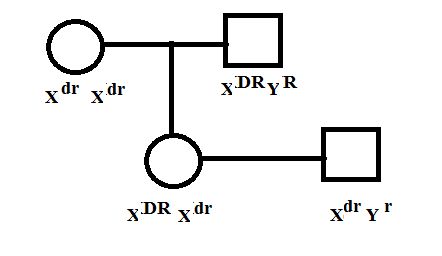
|
Mulher/Homem |
Xdr
(1/2) |
Yr
(1/2) |
|
XDR P |
XDR Xdr P(1/2) NR |
XDR Yr P(1/2) NR |
|
Xdr P |
Xdr Xdr P(1/2) DN |
Xdr Yr P(1/2) DN |
|
XDr
R |
XDr Xdr R(1/2) NN |
XDr Yr R(1/2) NN |
|
XdR
R |
XdR Xdr R(1/2) DR |
XdR Yr R(1/2) DR |
Sendo P = (1-d)/2 =(1 – 0,10)/2 = 0,45 = 45%
R = d/2 = 0,1/2 = 0,05 = 5%
(b) Qual
a proporção fenotípica esperada na descendência?
|
Mulheres |
|
|
Homens |
|
|
Normal, Retinite |
P/2 = 22,5% |
|
Normal, Retinite |
P/2 = 22,5% |
|
Daltonica, Normal |
P/2 = 22,5% |
|
Daltonica, Normal |
P/2 = 22,5% |
|
Normal, Normal |
R/2 = 2,5% |
|
Normal, Normal |
R/2 = 2,5% |
|
Daltonica, Retinite |
R/2 = 2,5% |
|
Daltonica, Retinite |
R/2 = 2,5% |
(c) Se o casal
deseja ter dois filhos, qual a probabilidade de ocorrer um menino normal e uma
menina normal, para as duas características?
|
Evento |
ni |
pi |
|
Menino normal, normal |
1 |
0,025 |
|
Menina normal, normal |
1 |
0,025 |
|
Outros |
0 |
0,95 |
|
Total |
2 |
1 |
P(Evento) = [N! / n1!
n2! N3!] (p1)n1 (p2)n2 (p3)n3
P(Evento) = [2! / 11!
1!] (0,025)1 (0,025)1 (0,95)0
14)
Considere cinco genes em nossa espécie, sendo dois autossomais (A/a e B/b), um
ligado ao sexo (C/c), um parcialmente ligado ao sexo (D/d) e um holândrico
(E/e). Responda:
(a) Represente seu genótipo, considerando que você é heterozigoto(a) ou
hemizigoto(a), dependendo do gene.
Mulher: Aa Bb XCD
Xcd ou Aa Bb XCd
XcD
Homem: Aa Bb XCD
YdE ou Aa Bb XCd
YDE Aa
Bb XCD
Yde ou Aa Bb XCd
YDe
Aa Bb XcD
YdE ou Aa Bb Xcd
YDE Aa
Bb XcD
Yde ou Aa Bb Xcd
YDe
(b) Para cada uma das situações a seguir, apresente os tipos e as probabilidades
dos óvulos ou espermatozoides que você produz. Em todos os casos, considere que
não há crossing-over na região entre os genes C/c e D/d nem na região de
homologia entre os cromossomos X e Y.
·
os dois genes
autossomais são independentes.
|
Mulher: Aa Bb
XCD Xcd |
|
Homem: Aa Bb
XCD YdE |
|
|
AB XCD |
(1/4)(1/4) P |
AB XCD |
(1/4)(1/4) P’ |
|
AB Xcd |
(1/4)(1/4) P |
AB YdE |
(1/4)(1/4) P’ |
|
AB XCd |
(1/4)(1/4) R |
AB XCd |
(1/4)(1/4) R’ |
|
AB XcD |
(1/4)(1/4) R |
AB YDE |
(1/4)(1/4) R’ |
|
|
|
|
|
|
Ab XCD |
(1/4)(1/4) P |
Ab XCD |
(1/4)(1/4) P’ |
|
Ab Xcd |
(1/4)(1/4) P |
Ab YdE |
(1/4)(1/4) P’ |
|
Ab XCd |
(1/4)(1/4) R |
Ab XCd |
(1/4)(1/4) R’ |
|
Ab XcD |
(1/4)(1/4) R |
Ab YDE |
(1/4)(1/4) R’ |
|
|
|
|
|
|
aB XCD |
(1/4)(1/4) P |
aB XCD |
(1/4)(1/4) P’ |
|
aB Xcd |
(1/4)(1/4) P |
aB YdE |
(1/4)(1/4) P’ |
|
aB XCd |
(1/4)(1/4) R |
aB XCd |
(1/4)(1/4) R’ |
|
aB XcD |
(1/4)(1/4) R |
aB YDE |
(1/4)(1/4) R’ |
|
|
|
|
|
|
ab XCD |
(1/4)(1/4) P |
ab XCD |
(1/4)(1/4) P’ |
|
ab Xcd |
(1/4)(1/4) P |
ab YdE |
(1/4)(1/4) P’ |
|
ab XCd |
(1/4)(1/4) R |
ab XCd |
(1/4)(1/4) R’ |
|
ab XcD |
(1/4)(1/4) R |
ab YDE |
(1/4)(1/4) R’ |
Sendo
P = (1-d)/2 R
= d/2 d= distância
entre os genes C/c e D/d
P’ = (1-d’)/2 R’
= d’/2 d’= distância
que representa a taxa de recombinação entre os cromossomos X e Y que envolve o
gene D/d
·
os dois genes
autossomais são completamente ligados – considere que eles estão em
aproximação.
Será considerado que os genes
estão em aproximação.
|
Mulher: Aa Bb
XCD Xcd |
|
Homem: Aa Bb
XCD YdE |
|
|
AB XCD |
(1/2)(1/2) P |
AB XCD |
(1/2)(1/2) P’ |
|
AB Xcd |
(1/2)(1/2) P |
AB YdE |
(1/2)(1/2) P’ |
|
AB XCd |
(1/2)(1/2) R |
AB XCd |
(1/2)(1/2) R’ |
|
AB XcD |
(1/2)(1/2) R |
AB YDE |
(1/2)(1/2) R’ |
|
|
|
|
|
|
|
|
|
|
|
ab XCD |
(1/2)(1/2) P |
ab XCD |
(1/2)(1/2) P’ |
|
ab Xcd |
(1/2)(1/2) P |
ab YdE |
(1/2)(1/2) P’ |
|
ab XCd |
(1/2)(1/2) R |
ab XCd |
(1/2)(1/2) R’ |
|
ab XcD |
(1/2)(1/2) R |
ab YDE |
(1/2)(1/2) R’ |
Sendo
P = (1-d)/2 R
= d/2 d= distância
entre os genes C/c e D/d
P’ = (1-d’)/2 R’
= d’/2 d’= distância
que representa a taxa de recombinação entre os cromossomos X e Y que envolve o
gene D/d
·
os dois genes
autossomais são ligados e a distância entre eles é 2 – considere que eles estão
em aproximação.
|
Mulher: Aa Bb
XCD Xcd |
|
Homem: Aa Bb
XCD YdE |
|
|
AB XCD |
P” P |
AB XCD |
P” P’ |
|
AB Xcd |
P” P |
AB YdE |
P” P’ |
|
AB XCd |
P” R |
AB XCd |
P” R’ |
|
AB XcD |
P” R |
AB YDE |
P” R’ |
|
|
|
|
|
|
Ab XCD |
R” P |
Ab XCD |
R” P’ |
|
Ab Xcd |
R” P |
Ab YdE |
R” P’ |
|
Ab XCd |
R” R |
Ab XCd |
R” R’ |
|
Ab XcD |
R” R |
Ab YDE |
R” R’ |
|
|
|
|
|
|
aB XCD |
R” P |
aB XCD |
R” P’ |
|
aB Xcd |
R” P |
aB YdE |
R” P’ |
|
aB XCd |
R” R |
aB XCd |
R” R’ |
|
aB XcD |
R” R |
aB YDE |
R” R’ |
|
|
|
|
|
|
ab XCD |
P” P |
ab XCD |
P” P’ |
|
ab Xcd |
P” P |
ab YdE |
P” P’ |
|
ab XCd |
P” R |
ab XCd |
P” R’ |
|
ab XcD |
P” R |
ab YDE |
P” R’ |
Sendo
P = (1-d)/2 R
= d/2 d= distância
entre os genes C/c e D/d
P’ = (1-d’)/2 R’
= d’/2 d’= distância
que representa a taxa de recombinação entre os cromossomos X e Y que envolve o
gene D/d
P” = (1-d”)/2 R
= d”/2 d” = distância
entre os genes C/c e D/d (no exemplo d” = 2cM = 0,02)
15) A doença de Huntington é causada por um
alelo letal dominante em homozigose (HH), localizado no autossomo 4. Nesse mesmo
cromossomo localiza-se o gene que determina o grupo sanguíneo MN. Um homem
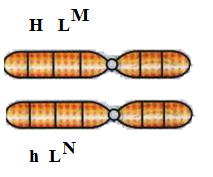
portador desse gene letal e com grupo sanguíneo MN (H LM/h LN)
casa-se com uma mulher de genótipo idêntico ao seu. Sabendo que os indivíduos
com grupo M e com grupo N são homozigotos (LMLM e LNLN, respectivamente),
responda:
|
|
Sugestão de leitura: http://www.minhavida.com.br/saude/temas/doenca-de-huntington http://www.abh.org.br/index.php?option=com_content&view=article&id=60&Itemid=58 |
|
|
|
(a) Quais as
proporções genotípica e fenotípica esperadas na descendência, admitindo que os
genes sejam completamente ligados?
|
Homem:
|
X |
Mulher:
|
Faremos o cruzamento agora para encontras a descendência:
|
Homem/Mulher |
HLM |
hLN |
|
HLM |
HH
LMLM
- Inviável (*) |
Hh LMLN |
|
hLN |
Hh LMLN |
hh LNLN |
-Proporção genotípica:
2\3 Hh LMLN
1\3 hh LNLN
-Proporção fenotípica:
2\3 Portador de Huntington,
grupo MN
1\3 Normal, Grupo N
(b) Quais as
proporções genotípica e fenotípica esperadas na descendência, admitindo que os genes
tenham distribuição independente?
Resposta por meio da análise conjunta:
|
Homem/Mulher |
H LM |
h LN |
H LN |
h LM |
|
HLM |
HH
LMLM
- Inviável (*) |
Hh LMLN “Norm,MN” |
HH
LMLN
- Inviável (*) |
Hh LMLN “Norm,MN” |
|
h LN |
Hh LMLN “Norm,M” |
hh LNLN “Norm,NN” |
Hh LNLN “Norm,N” |
hh LMLN “Norm,MN” |
|
H LN |
HH
LMLN - Inviável (*) |
Hh LNLN “Norm,N” |
HH
LNLN
- Inviável (*) |
Hh LMLN
“Norm,MN” |
|
h LM |
Hh LMLM “Norm,M” |
hh LMLN “Norm,MN” |
Hh LMLN “Norm,MN” |
hh LMLM “Norm,M” |
Resposta por meio da análise separada:
|
Gametas |
LM |
LN |
|
LM |
LM LM |
LM LN |
|
LN |
LM LN |
LN LN |
|
Gametas |
H |
h |
|
H |
H H |
H h |
|
h |
H h |
h h |
Conclusão:
|
Fenótipo |
Genótipo |
Frequencia Genotípica |
Frequencia fenotípica |
|
Normal, M |
Hh LM LM
hh LM LM |
(2/3)x(1/4) = 2/12 (1/3)x(1/4) = 1/12 |
3/12 |
|
Normal, N |
Hh LN LN hh LN LN |
(2/3)x(1/4) = 2/12 (1/3)x(1/4) = 1/12 |
3/12 |
|
Normal, MN |
Hh LM LN
hh LM LN |
(2/3)x(2/4) = 4/12 (1/3)x(2/4) = 2/12 |
6/12 |
© Admita que os
dois genes estejam separados por seis unidades de mapa e apresente as
proporções genotípicas e fenotípicas esperadas na descendência (na espécie
humana ocorre crossing-over nos dois sexos).
|
Homem/Mulher |
H LM P |
h LN P |
H LN R |
h LM R |
|
HLM P |
HH
LMLM
- Inviável (*) |
Hh LMLN “Norm,MN” |
HH
LMLN
- Inviável (*) |
Hh LMLN “Norm,MN” |
|
h LN P |
Hh LMLN “Norm,M” |
hh LNLN “Norm,NN” |
Hh LNLN “Norm,N” |
hh LMLN “Norm,MN” |
|
H LN R |
HH
LMLN - Inviável (*) |
Hh LNLN “Norm,N” |
HH
LNLN
- Inviável (*) |
Hh LMLN “Norm,MN” |
|
h LM R |
Hh LMLM “Norm,M” |
hh LMLN “Norm,MN” |
Hh LMLN “Norm,MN” |
hh LMLM “Norm,M” |
|
Fenótipo |
Genótipo |
Frequencia Genotípica |
Frequencia fenotípica |
Frequencia fenotípica (sem letal) |
|
Normal, M |
Hh LM LM
hh LM LM |
P2 +PR R2 |
P2 +PR+ R2 =
0.2359 |
0.3145 |
|
Normal, N |
Hh LN LN hh LN LN |
P2 +PR PR |
P2
+2PR = 0.2491 |
0.3321 |
|
Normal, MN |
Hh LM LN
hh LM LN |
P2 + PR +
2 R2 2PR |
P2 +3PR+
2R2 = 0.265 |
0.3533 |
|
Total |
|
|
0.75 |
1,0 |
Letal: P2
+2PR+ R2 = 0,25
Sendo
P = (1-d)/2 = (1- 0,06)/2 =0,47
R = d/2 = 0,06/2 = 0,03
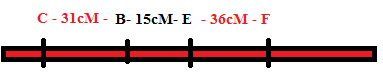
16) Na matriz a seguir estão as porcentagens de
gametas recombinantes em relação a seis genes de uma dada espécie. Analise e
represente o mapa de ligação, com a ordem correta dos genes em cada grupo.

Formação dos grupos de ligação 1
|
Passo
1. Genes mais próximos: A e D |
|
|
Passo
2: Mais próximos às extremidades: De
A: B, C,E,F todos independentes De
D: B,C,E,F todos independentes |
|
Formação dos grupos de ligação 2
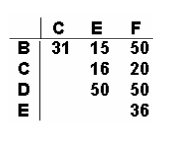
|
Passo
1. Genes mais próximos: B e E |
|
|
Passo
2: Mais próximos às extremidades: De
B: C com 31cM De
E: F com 36cM |
|
17) Em Drosophila
melanogaster, os fenótipos mutantes 'yellow' (corpo amarelo), 'scute'
(cerdas em escudo) e 'white' (olhos brancos) são
determinados por genes recessivos ligados ao sexo (ao X). Fêmeas triplo-heterozigotas,
selvagens, foram cruzadas com machos com corpo amarelo, cerdas em escudo e
olhos vermelhos (hemizigotos), produzindo os
resultados a seguir. Responda:
F1: 425 fêmeas e machos com corpo amarelo, cerdas em escudo e
olhos brancos
7 fêmeas e machos com corpo
amarelo, cerdas em escudo e olhos vermelhos
429 fêmeas e machos com corpo cinza, cerdas normais e olhos vermelhos
6 fêmeas e machos com corpo
cinza, cerdas normais e olhos brancos
|
Corpo |
|
Cerdas |
|
Olhos |
|
|
Cinza |
|
Normal |
|
Vermelho |
|
|
Amarelo |
|
Escudo |
|
Branco |
|
(a) Qual a distância entre os locos 'yellow'
e 'scute'? Qual a frequência observada de crossing-over
entre esses dois locos?
Faremos a análise das fêmeas pois
em macho de drosophila não corre permuta. Assim, temos o cruzamento:
Yy
Ss Ww x yy ss ww
|
Corpo |
Cerdas |
Olhos |
Observado |
|
Cinza |
Normal |
Vermelho |
429 |
|
Cinza |
Normal |
Brancos |
6 |
|
Cinza |
Escudo |
Vermelho |
|
|
Cinza |
Escudo |
Brancos |
|
|
|
|
|
|
|
Amarelo |
Normal |
Vermelho |
|
|
Amarelo |
Normal |
Brancos |
|
|
Amarelo |
Escudo |
Vermelho |
7 |
|
Amarelo |
Escudo |
Brancos |
425 |
Fazendo análise dos genes Y/y
e S/s temos:
|
Corpo |
Cerdas |
Observado |
Esperado
Ho: genes independentes |
Esperado.
Ho: genes ligados |
|
Cinza |
Normal |
429+6
=435 |
(1/4)867=216,75 |
P |
|
Cinza |
Escudo |
0 |
(1/4)867=216,75 |
R |
|
|
|
|
|
|
|
Amarelo |
Normal |
0 |
(1/4)867=216,75 |
R |
|
Amarelo |
Escudo |
425+7 = 432 |
(1/4)867=216,75 |
P |
|
Total |
|
867 |
|
|
Sempre quando for ‘yellow’ é ‘scute’, desta maneira
estão completamente ligados, ou seja, estão na mesma posição.
Portanto não há crossing-over e a distância é zero.
(b) Qual a distância entre os locos 'yellow'
e 'white'? A frequência de
crossing-over entre esses dois locos é menor, igual ou superior à frequência de
crossing-over entre os locos 'yellow' e 'scute'?
Fazendo análise dos genes Y/y
e W/w temos:
|
Corpo |
Olhos |
Observado |
Esperado
Ho: genes independentes |
Esperado.
Ho: genes ligados |
|
Cinza |
Vermelho |
429 |
(1/4)867=216,75 |
P |
|
Cinza |
Brancos |
6 |
(1/4)867=216,75 |
R |
|
|
|
|
|
|
|
Amarelo |
Vermelho |
7 |
(1/4)867=216,75 |
R |
|
Amarelo |
Brancos |
425 |
(1/4)867=216,75 |
P |
|
Total |
|
867 |
|
|
Como o desvio entre observado e
esperado, para a hipótese de que os genes são independentes, rejeitamos Ho: Y/y e W/w independentes.
Há, portanto, evidências de
ligação fatorial, ou seja:
d(Y/y-W/w)= [(R + R)100]/Total = [(7+6)
100]/867 = 1,49 cM
(c) Apresente as
proporções genotípicas e fenotípicas esperadas nas gerações F1 e F2 do cruzamento
entre fêmeas com corpo amarelo e cerdas em escudo e machos com corpo cinza e
cerdas normais.
Para iniciar faremos o genótipo dos indivíduos do cruzamento:
Cruzamento: XysXys
x XYSY
Faremos a tabela contendo o resultado dos cruzamentos:
Geração F1:
|
Fêmea/Macho |
XYS |
Y |
|
Xys |
XYS Xys corpo cinza e cerdas normais. |
XYSY corpo cinza e cerdas normais. |
Portanto a F1 possui a seguinte proporção:
F1: 1\2 XYS Xys
: 1\2 XYSY
Geração F2:
|
Fêmea/Macho |
XYS |
Y |
|
XYS |
XYS XYS |
XYSY |
|
Xys |
XYS Xys |
XysY |
(*) Não formam gametas recombinantes pois
os genes estão completamente ligados
F2: 1\4 XYS XYS :
1\4 XYSY : 1\4 XYSXys : 1\4 XysY
18) Considere que os resultados a seguir
(hipotéticos) foram obtidos em um estudo visando à identificação de marcadores
RAPD para o gene de resistência do cultivar de feijoeiro comum Perry Marrow à raça alfa de Colletotrichum lindemuthianum,
agente causal da antracnose.
P:
Perry Marrow (P1), resistente, com os
marcadores 1 e 2 x Michelite
(P2), suscetível, sem os marcadores 1 e 2
F1: 100%
de plantas resistentes, com os marcadores 1 e 2
Cruzamaneto: F1 x P2
F1: 63
plantas resistentes, com os marcadores 1 e 2
10
plantas resistentes, com o marcador 1 e sem o marcador
2
14
plantas suscetíveis, sem o marcador 1 e com o marcador
2
61
plantas suscetíveis, sem os marcadores 1 e 2
Responda:
(a) Os dois marcadores são ligados ao gene de resistência? Justifique
sua resposta.
|
Marcador 1 |
Marcador 2 |
Resistência |
Observado |
|
+ |
+ |
R |
63 |
|
+ |
+ |
S |
|
|
+ |
- |
R |
10 |
|
+ |
- |
S |
|
|
|
|
|
|
|
- |
+ |
R |
|
|
- |
+ |
S |
14 |
|
- |
- |
R |
|
|
- |
- |
S |
61 |
Análise
do marcador 1 e o gene da resistência
|
Marcador 1 |
Resistência |
Observado |
Esperado. Ho: independentes |
Esperado. Ho: ligados |
|
+ |
R |
63+10=73 |
(1/4)148
=37 |
P |
|
+ |
S |
0 |
(1/4)148
=37 |
R |
|
- |
R |
0 |
(1/4)148
=37 |
R |
|
- |
S |
61+14=75 |
(1/4)148
=37 |
P |
|
Total |
|
148 |
|
|
Conclusão: Os desvios entre observado e esperado são grandes indicando a rejeição da hipótese de independência. Há evidência de exista ligação fatorial, porém como não ocorrem classes recombinantes a ligação deve ser completa.
Análise
do marcador 2e o gene da resistência
|
Marcador 2 |
Resistência |
Observado |
Esperado. Ho: independentes |
Esperado. Ho: ligados |
|
+ |
R |
63 |
(1/4)148
=37 |
P |
|
+ |
S |
14 |
(1/4)148
=37 |
R |
|
- |
R |
10 |
(1/4)148
=37 |
R |
|
- |
S |
61 |
(1/4)148
=37 |
P |
|
Total |
|
148 |
|
|
Conclusão: Os desvios entre observado e esperado são grandes indicando a rejeição da hipótese de independência. Há evidência de exista ligação fatorial e a distância é dada por:
D = [(R + R) 100]/Total [14 + 10)100]/148 = 16,2cM
(b) Se o
marcador 1 é ligado ao gene de resistência, calcule a
distância entre eles.
Distância= 0% de recombinação
Isto significa que o marcador
1 está completamente ligado ao gene de resistência,
desta maneira podemos observar que todos os indivíduos que manifestam a
resistência apresentam também presença do marcador 1.
(c) Se o
marcador 2 é ligado ao gene de resistência, calcule a
distância entre eles.
Como já demonstrado:
d= 16,2
cM
(d) Se ambos
marcam o gene de resistência, qual o melhor? Explique sua resposta.
Marcador 1, pois ele se encontra exatamente na mesma posição em que o gene de
resistência se encontra.
19) Em Drosophila
melanogaster, os fenótipos mutantes com veias
nítidas, antenas sem aresta e corpo manchado são determinados por genes
recessivos autossomais (n, a e m, respectivamente). Os alelos
dominantes desses genes determinam, respectivamente, veias normais, antenas com
aresta e corpo sem manchas. Considere os resultados do seguinte
cruzamento-teste e responda:

P:
fêmea c/asas c/veias normais c/antenas com aresta e de corpo sem manchas x
macho c/asas c/veias nítidas, c/antenas sem aresta e de corpo manchado
F1: 89
moscas c/veias normais, antenas s/aresta, corpo s/manchas
90 moscas
c/veias normais, antenas s/aresta, corpo manchado
89
moscas c/veias nítidas, antenas c/aresta, corpo s/manchas
88
moscas c/veias nítidas, antenas c/aresta, corpo manchado
(a) Qual sua conclusão a respeito da localização dos genes N/n e A/a?
Explique sua resposta. Se forem ligados, calcule a distância entre eles.
|
Veias (N/n) |
Antenas (A/a) |
Corpo (M/m) |
Observado |
|
Normal |
S/ aresta |
S/ manchas |
89 |
|
Normal |
S/ aresta |
manchado |
90 |
|
Normal |
C/ aresta |
S/ manchas |
|
|
Normal |
C/ aresta |
manchado |
|
|
|
|
|
|
|
Nítidas |
S/ aresta |
S/ manchas |
|
|
Nítidas |
S/ aresta |
manchado |
|
|
Nítidas |
C/ aresta |
S/ manchas |
89 |
|
Nítidas |
C/ aresta |
manchado |
88 |
Análise dos genes N/n e A/a
|
Veias (N/n) |
Antenas (A/a) |
Observado |
Esperado. Genes independentes |
Esperado. Genes ligados |
|
Normal |
S/ aresta |
89+90=
179 |
(1/4)356=89 |
P |
|
Normal |
C/ aresta |
0 |
(1/4)356=89 |
R |
|
Nítidas |
S/ aresta |
0 |
(1/4)356=89 |
R |
|
Nítidas |
C/ aresta |
89+88 =
177 |
(1/4)356=89 |
P |
|
Total |
|
356 |
|
|
Conclusão: Os desvios entre observado e esperado são grandes indicando a rejeição da hipótese de independência. Há evidência de exista ligação fatorial, porém como não ocorrem classes recombinantes a ligação deve ser completa.
(b) Qual
sua conclusão a respeito da localização dos genes N/n e M/m? Explique sua
resposta. Se forem ligados, calcule a distância entre eles.
Análise dos genes N/n e M/m
|
Veias (N/n) |
Corpo (M/m) |
Observado |
Esperado. Genes independentes |
|
Normal |
S/ manchas |
89 |
(1/4)356=89 |
|
Normal |
manchado |
90 |
(1/4)356=89 |
|
Nítidas |
S/ manchas |
89 |
(1/4)356=89 |
|
Nítidas |
manchado |
88 |
(1/4)356=89 |
|
Total |
|
356 |
|
Conclusão: Os desvios entre
observado e esperado são pequenos indicando a não rejeição da hipótese
de independência. Assim, considera-se que os genes N/n e M/m pertencem a cromossomos
diferentes (não ligados)
(c)
Apresente as proporções genotípica e fenotípica esperadas na descendência do
cruzamento entre indivíduos com veias normais e antenas com aresta, com
genótipos idênticos aos da fêmea usada no cruzamento-teste.
O cruzamento realizado é NnAa x nnaa
Como os genes estão
completamente ligados, logo não temos recombinantes.
P: NnAa = ½ (Veias normais e com aresta)
P: nnaa
= ½ (Veias nítidas e sem aresta)
20) Em Drosophila melanogaster,
os fenótipos mutantes brown (olhos marrom), ebony (corpo preto), curved (asas
curvadas) e narrow (asas estreitas) são determinados
por genes recessivos autossomais (b, e, c, n,
respectivamente). Seus respectivos alelos determinam olhos vermelhos, corpo
cinza, asas não curvadas e asas não estreitas. Analise os resultados dos
seguintes cruzamentos e responda:
|
Corpo |
|
Asas |
|
Olhos |
|
Asas |
|
|
Cinza |
|
Normal |
|
Vermelho |
|
Normal |
|
|
Preto |
|
Estreitas |
|
Marrom |
|
Curvada |
|
Cruzamento 1:
P: fêmea com asas normais x macho com asas curvadas e estreitas
F1: 35 moscas com asas normais
187 moscas com asas curvadas
190 moscas com asas estreitas
31 moscas com asas curvadas e estreitas
Cruzamento 2:
P: fêmea com olhos vermelhos e corpo cinza x macho com olhos
marrons e corpo preto
F1: 127 moscas com olhos vermelhos e corpo cinza
125 moscas com olhos vermelhos e corpo preto
126 moscas com olhos marrons e corpo cinza
123 moscas com olhos marrons e corpo preto
(a) É possível afirmar que os genes c e n são ligados? Justifique
sua resposta. Em caso positivo, calcule a distância entre eles. Resp.: 14,89 cM.
|
Cruzamento 1 |
|
|
|
Cruzamento 2 |
|
|
|
Asa Normal/Curvada C/c |
Asa Normal/Estreita N/n |
Observado |
|
Olho Vermelho/marrom B/b |
Corpo Cinza/Preto E/e |
Observado |
|
Normal |
Normal |
35 |
|
Vermelho |
Cinza |
127 |
|
Normal |
Estreita |
190 |
|
Vermelho |
Preto |
125 |
|
|
|
|
|
|
|
|
|
Curvada |
Normal |
187 |
|
Marrom |
Cinza |
126 |
|
Curvada |
Estreita |
31 |
|
Marrom |
Preto |
123 |
Análise dos genes
C/c e N/n
Inicialmente consideraremos a hipótese dos genes serem
independentes. Ho: C/c
e N/n não ligados
|
Cruzamento 1 |
|
|
|
|
|
|
Asa Normal/Curvada C/c |
Asa Normal/Estreita N/n |
Observado |
Esperado. Ho: genes
independentes |
(O – E)2/E |
Tipo de gametas (Considerando
genes ligados) |
|
Normal |
Normal |
35 |
(1/4)443 =110,75 |
51,81 |
R |
|
Normal |
Estreita |
190 |
(1/4)443 =110,75 |
52,49 |
P |
|
|
|
|
|
|
|
|
Curvada |
Normal |
187 |
(1/4)443 =110,75 |
56,70 |
P |
|
Curvada |
Estreita |
31 |
(1/4)443 =110,75 |
57,42 |
R |
|
Total |
|
443 |
|
218,42 |
|
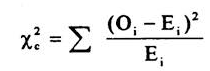
Logo
X2 = 218,42
GL = 4 -1 = 3.
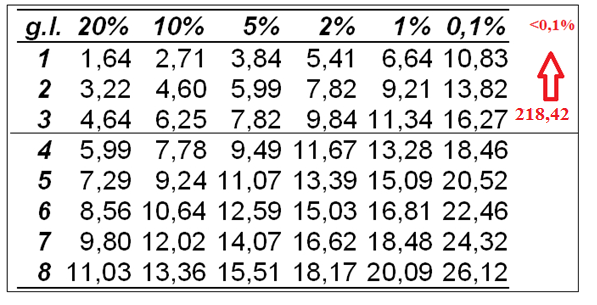
Conclusão: O valor de a < 0,001 indicado baixo erro ao rejeitar Ho.
Portanto, há evidências de que os genes estão ligados.
A distância entre os genes é dada por:
d(C/c-N/n)=[100(R + R)]/Total =
[100(35+31]/443= 14,85 cM
(b) É
possível que os genes b e e
sejam ligados? Justifique sua resposta. Em caso positivo, calcule a distância
entre eles.
Para esse caso
teremos que fazer uma análise de qui-quadrado, com o
cruzamento:
Cruzamento: BbEe x bbee
|
Cruzamento 2 |
|
|
|
|
|
Olho Vermelho/marrom B/b |
Corpo Cinza/Preto E/e |
Observado |
Esperado. Ho: genes
independentes |
(O – E)2/E |
|
Vermelho |
Cinza |
127 |
(1/4)501 = 125.25 |
0,017 |
|
Vermelho |
Preto |
125 |
(1/4)501 = 125.25 |
0,0005 |
|
|
|
|
|
|
|
Marrom |
Cinza |
126 |
(1/4)501 = 125.25 |
0,004 |
|
Marrom |
Preto |
123 |
(1/4)501 = 125.25 |
0,004 |
|
Cruzamento 2 |
|
501 |
|
0,025 |
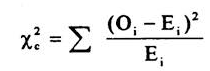
Logo
X2 = 0,025
GL = 4 -1 = 3.
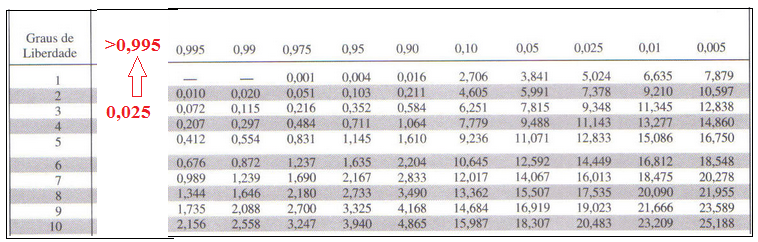
Conclusão: O valor de a >
0,995 indicado alto erro
ao rejeitar Ho. Portanto, concluímos que os genes são independentes e a
hipótese Ho: a segregação ocorre 1:1:1:1 é aceitável.
.
21) Em Drosophila
melanogaster, os genes recessivos autossomais b e p determinam corpo preto e olhos púrpura,
respectivamente. Seus alelos dominantes B e P condicionam corpo cinza e olhos
vermelhos, respectivamente. A distância entre os locos B/b e P/p é de 6 unidades de mapa. Com base nessas informações, responda:
|
Corpo |
|
Olhos |
|
|
Cinza |
|
Vermelho |
|
|
Preto |
|
Púrpura |
|
(a) Quais as proporções genotípicas e
fenotípicas esperadas na descendência do cruzamento entre fêmea tipo selvagem
duplo-heterozigota em fase de aproximação (BP/bp) e macho de corpo preto e olhos púrpura?
Nesse caso temos os seguintes genótipos para o cruzamento:
Cruzamento: fêmea (BP/bp) x
macho (bp/bp)
|
Fêmea/Macho |
bp |
Fenótipo |
Frequencia |
|
BP P |
BP // bp |
Cinza,
Vermelho |
P =
0,47 |
|
bp
P |
bp // bp |
Preto,
Púrpura |
P =
0,47 |
|
Bp
R |
Bp // bp |
Cinza,
Púrpura |
R =
0,03 |
|
bP
R |
bP // bp |
Preto,
Vermelho |
R =
0,03 |
Sendo:
P = (1-d)/2=
(1-0,06)/2 = 0,47
R = d/2 = 0,06/2 = 0,03
(b) Quais as proporções
genotípicas e fenotípicas esperadas na descendência do cruzamento de uma fêmea
tipo selvagem duplo-heterozigota em fase de repulsão (Bp/bP) com um macho de
corpo preto e olhos púrpura?
Mesmo modo de pensar da letra a:
Cruzamento: fêmea (Bp/bP) x macho (bp/bp)
|
Fêmea/Macho |
bp |
Fenótipo |
Frequencia |
|
Bp
P |
Bp // bp |
Cinza,
Púrpura |
P =
0,47 |
|
bP
P |
bP // bp |
Preto,
Vermelho |
P =
0,47 |
|
BP R |
BP // bp |
Cinza,
Vermelho |
R =
0,03 |
|
bp
R |
bp // bp |
Preto,
Púrpura |
R =
0,03 |
Sendo:
P = (1-d)/2=
(1-0,06)/2 = 0,47
R = d/2 = 0,06/2 = 0,03
(c) Quais as proporções
genotípicas e fenotípicas esperadas na descendência do cruzamento de uma fêmea
de corpo preto e olhos púrpuras com um macho duplo-heterozigoto em
fase de aproximação? (admita que nos machos não ocorre
crossing-over).
Nesse caso temos os seguintes genótipos para o cruzamento:
Cruzamento: macho (BP/bp) x fêmea (bp/bp)
-Gametas:

Uma tabela para os cruzamentos:
|
Fêmea/Macho |
Bp ( ½ ) |
Bp ( ½ ) |
|
bp |
Bp
bp |
bp
bp |
Com isso temos:
-Proporção fenotípica:
Corpo cinza e olhos vermelhos (BbPp): 0,5
Corpo preto e olhos púrpura (bbpp): 0,5
22) Em Drosophila melanogaster,
os fenótipos mutantes taxi (asas abertas) e ebony (corpo
preto) são determinados por genes recessivos. Analise os resultados
apresentados a seguir, obtidos pelos alunos de BIO241 - Laboratório de Genética
Bási e responda:
Cruzamento 1:
P: fêmea selvagem, duplo-heterozigota x macho taxi, ebony
F1: 226 moscas selvagens
41 moscas ebony
34 moscas taxi
232 moscas taxi, ebony
Cruzamento 2:
P: fêmea taxi, ebony x macho selvagem,
duplo-heterozigoto
F1: 60 moscas selvagens
0 mosca ebony
0 mosca
taxi
40 moscas taxi, ebony
|
Corpo |
|
Asas |
|
|
Selvagem |
|
Selvagem |
|
|
Ebony |
|
Taxi |
|
(a) Os dois genes são ligados? Justifique sua resposta.
|
Cruzamento 1 |
TeEe x ttee |
|
|
Cruzamento 2 |
ttee x TeEe |
|
|
Asas |
Corpo |
Observado |
|
Asas |
Corpo |
Observado |
|
Selvagem |
Selvagem |
226 |
|
Selvagem |
Selvagem |
60 |
|
Selvagem |
Ebony |
41 |
|
Selvagem |
Ebony |
0 |
|
Taxi |
Selvagem |
34 |
|
Taxi |
Selvagem |
0 |
|
Taxi |
Ebony |
232 |
|
Taxi |
Ebony |
40 |
Cruzamento 1: TeEe (fêmea)
x ttee (macho)
E a hipótese testada é de:
Ho: A segregação dos genes ocorre 1:1:1:1
|
Gametas Fêmea \Macho |
te |
Fenótipo |
Observado |
Esperado (Ho.: genes independentes) |
(O – E)2/E |
Tipo de gameta da fêmea (Ho: genes ligados) |
|
TE |
TtEe |
Selvagem |
226 |
(1/4)533 = 133.25 |
74,03 |
P |
|
Te |
Ttee |
Ebony |
41 |
(1/4)533 = 133.25 |
66,13 |
R |
|
tE |
ttEe |
Taxi |
34 |
(1/4)533 = 133.25 |
76,26 |
R |
|
te |
ttee |
Taxi, Ebony |
232 |
(1/4)533 = 133.25 |
68,24 |
P |
|
Total |
|
|
533 |
|
284,67 |
|
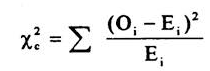
Logo
X2 = 284,67
GL = 4 -1 = 3.
Consultando uma tabela apropriada constatamos
que a < 0,01%. Logo é pequeno o
erro ao rejeitar a hipótese e independência e concluímos que há evidências de
ligação fatorial.
A distância entre os genes deverá ser de:
d =[100(R + R)]/Total = [100(41+34]/533=
14,07 cM
Cruzamento 2: TeEe (macho) x ttee (fêmea)
E a hipótese testada é de:
Ho: A segregação dos genes ocorre 1:1:1:1
|
Gametas Macho \Fêmaa |
te |
Fenótipo |
Observado |
Esperado (Ho.: genes independentes) |
|
TE |
TtEe |
Selvagem |
60 |
(1/4)100 = 25 |
|
Te |
Ttee |
Ebony |
|
(1/4)100 = 25 |
|
tE |
ttEe |
Taxi |
|
(1/4)100 = 25 |
|
te |
ttee |
Taxi, Ebony |
40 |
(1/4)100 = 25 |
|
Total |
|
|
100 |
|
Constamos a falta de algumas
classes que seriam esperadas para os genes independentes. Tal fato é indicativo
de que os genes
estão ligados e, pelos resultados observados, é fácil concluir que o macho é um
duplo-heterozigoto em aproximação (TE // te). Como não há permuta os machos
produzirão apenas dois tipos de gametas com frequência igual a 50%, ou seja:
|
Gametas Macho \Fêmaa |
te |
Fenótipo |
Observado |
Esperado (Ho.: genes independentes) |
|
TE |
TtEe |
Selvagem |
60 |
(1/2)100 = 50 |
|
te |
ttee |
Taxi, Ebony |
40 |
(1/2)100 = 50 |
|
Total |
|
|
100 |
|
(b) Se os genes são ligados, ocorre crossing-over nas fêmeas e nos
machos? Justifique sua resposta.
- O
cruzamento 1 permitiu concluir que a hipótese de que
os genes são ligados era falsa. Assim, foi evidenciada a ligação fatorial e por
meio da frequência dos gametas recombinantes foi estimada a distância em 14,07 cM. A ocorrência de classes
gaméticas recombinantes é evidência de que a permuta ocorre nas fêmeas.
- O
cruzamento 2 também permite evidenciar que a hipótese
de independência entre os genes é falsa. Este cruzamento não permite estimar a
distância entre o genes, mas ratifica o fato da
ligação fatorial associado ao fenômeno de ausência de permuta nos machos de drosophila,
23) No caupi, o alelo A, dominante, é responsável pela resistência
ao vírus da mancha anelar, e o alelo B, também dominante, confere resistência
ao vírus do mosaico. O cruzamento de uma planta resistente às duas doenças com
uma suscetível produziu a seguinte descendência: 145 plantas resistentes aos
dois vírus, 151 plantas suscetíveis aos dois vírus, 450 plantas resistentes
apenas ao vírus da mancha anelar e 440 plantas resistentes apenas ao vírus do
mosaico.
a) Os genes são
ligados? Justifique.
Incialmente será testada a hipótese que os
genes são independentes:
Cruzamento: AaBb x aabb
|
Gametas |
a b |
|
Mancha anelar |
Mosaico |
Observado |
Esperado (Ho: genes A/a e B/b independentes) |
(O – E)2/E |
|
A
B |
Aa Bb |
|
Resistente |
Resistente |
145 |
(1/4) 1186 = 296,5 |
77,41 |
|
A b |
Aa bb |
|
Resistente |
Suscetível |
450 |
(1/4) 1186 = 296,5 |
79,47 |
|
a B |
aa Bb |
|
Suscetível |
Resistente |
440 |
(1/4) 1186 = 296,5 |
69,45 |
|
a b |
aa bb |
|
Suscetível |
Suscetível |
151 |
(1/4) 1186 = 296,5 |
71,40 |
|
Total |
|
|
|
|
1186 |
|
297,73 |
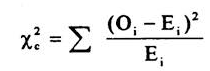
Logo
X2 = 297,73
GL = 4 -1 = 3.
Consultando uma tabela apropriada constatamos
que a < 0,01%. Logo é pequeno o
erro ao rejeitar a hipótese e independência e concluímos que há evidências de
ligação fatorial.
b) Qual é a
distância entre os dois genes?
Para isto deve-se
identificar as classes gaméticas paternais (mais frequentes) e recombinantes
(menos frequentes)
|
Gametas |
a b |
|
Mancha anelar |
Mosaico |
Observado |
Classificação dos gametas do duplo-heterozigoto |
|
A
B |
Aa Bb |
|
Resistente |
Resistente |
145 |
R |
|
A b |
Aa bb |
|
Resistente |
Suscetível |
450 |
P |
|
a B |
aa Bb |
|
Suscetível |
Resistente |
440 |
P |
|
a b |
aa bb |
|
Suscetível |
Suscetível |
151 |
R |
|
Total |
|
|
|
|
1186 |
|
A distância é calcula pela mesma fórmula:
d(A/a-B/b)= [100(R + R) ] /Total = [100(151+145)]/1186 = 24,96 cM
c) Atribuir
genótipos
Genitores: Resistente A b // a B x suscetível a b // a b
Descendência
|
Gametas |
a b |
|
Mancha anelar |
Mosaico |
|
A
B |
A B // a b |
|
Resistente |
Resistente |
|
A b |
A b // a b |
|
Resistente |
Suscetível |
|
a B |
a B // a b |
|
Suscetível |
Resistente |
|
a b |
a b // a b |
|
Suscetível |
Suscetível |
Os indivíduos terão a seguinte configuração no
cromossomo: Ab//ab
d) Qual é a fase de
ligação dos genes no progenitor resistente?
É reconstituído por meio das informações de seus gametas partenais. Assim, a fase é de repulsão (A b // a B)
24) No caupi, os genes A/a e B/b conferem resistência a dois
vírus, conforme explicado no exercício anterior, e estão localizados num mesmo
cromossomo, a 25 cM de distância. Uma variedade
(homozigota) resistente aos dois vírus foi cruzada com uma variedade
suscetível. A geração F1 resultante foi submetida a um cruzamento-teste,
produzindo 2.000 descendentes. Qual é o número esperado de cada fenótipo?

Genitores: Resistente: A B
// A B x Suscetível: a b / / a b
Geração F1: A B / / a b - Resistente
Cruzamento teste: F1: A B / / a b x Testador: a b / / a b
Resultado:
|
Gametas F1 \ Testador |
a b |
|
Mancha anelar |
Mosaico |
Frequencia |
Esperado em 2000 descendentes |
|
A
B |
Aa Bb |
|
Resistente |
Resistente |
P = (1-d)/2 = (1-0,25)/2 = 0,375 |
0,375(2000) = 750 |
|
A b |
Aa bb |
|
Resistente |
Suscetível |
R = d/2 = 0,25/2 = 0,125 |
0,125(2000) = 250 |
|
a B |
aa Bb |
|
Suscetível |
Resistente |
R = d/2 = 0,25/2 = 0,125 |
0,125(2000) = 250 |
|
a b |
aa bb |
|
Suscetível |
Suscetível |
P = (1-d)/2 = (1-0,25)/2 = 0,375 |
0,375(2000) = 750 |
25) Pericarpo vermelho
no milho é devido ao alelo dominante I, enquanto pericarpo incolor é
condicionado pelo alelo recessivo i. Sementes no pendão são devidas ao alelo
recessivo s, enquanto pendão normal depende do alelo S. O cruzamento-teste de
duas plantas produziu a seguinte descendência:
|
|
|
|

a) Quais são os genótipos das plantas 1 e 2?
Análise da planta 1 – P1
- Análise do
pericarpo
|
Pericarpo |
Observado |
Conclusão: |
|
Vermelho |
15+1219 = 1234 |
Padrão de Segregação : 1: 1 |
|
Incolor |
14+1174= 1188 |
Cruzamento: Ii x ii |
- Análise do
pendão
|
Pendão |
Observado |
Conclusão: |
|
Normal |
14+1219 = 1233 |
Padrão de Segregação : 1: 1 |
|
Com sementes |
15+1174 = 1189 |
Cruzamento: Ss
x ss |
- Análise conjunta
para detecção de independência ou ligação fatorial
|
Pericarpo |
Pendão |
Observado |
Esperado. Ho: genes
independentes |
(O – E)2/E |
Tipo de gameta da P1 |
|
Vermelho |
Normal |
1219 |
( ¼ )2422 = 605,5 |
621,60 |
I S (P) |
|
Vermelho |
Com sementes |
15 |
( ¼ )2422 = 605,5 |
575,87 |
I s (R) |
|
Incolor |
Normal |
14 |
( ¼ )2422 = 605,5 |
577,82 |
i S (R) |
|
Incolor |
Com sementes |
1174 |
( ¼ )2422 = 605,5 |
533,76 |
i s (P) |
|
Total |
|
2422 |
|
2309,06 |
|
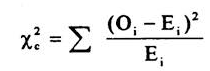 Logo X2 = 2309,06 GL = 4 -1 = 3.
Logo X2 = 2309,06 GL = 4 -1 = 3.
Consultando uma tabela
apropriada constatamos que a <
0,01%. Logo é pequeno o erro ao rejeitar a hipótese e independência e concluímos
que há evidências de ligação fatorial. O genótipo de P1 pode ser estabelecido a
partir dos gametas paternais (mais frequentes). Assim, concluímos que: P1 = I S // i s
Análise da planta 2 – P2
- Análise do
pericarpo
|
Pericarpo |
Observado |
Conclusão: |
|
Vermelho |
580 + 9 = 589 |
Padrão de Segregação : 1: 1 |
|
Incolor |
610 + 8 = 618 |
Cruzamento: Ii x ii |
- Análise do
pendão
|
Pendão |
Observado |
Conclusão: |
|
Normal |
610 + 9 = 619 |
Padrão de Segregação : 1: 1 |
|
Com sementes |
580 + 8 = 588 |
Cruzamento: Ss
x ss |
- Análise
conjunta para detecção de independência ou ligação fatorial
|
Pericarpo |
Pendão |
Observado |
Esperado. Ho: genes
independentes |
(O – E)2/E |
Tipo de gameta da P2 |
|
Vermelho |
Normal |
9 |
( ¼ )1207 = 301.75 |
284,02 |
I S (R) |
|
Vermelho |
Com sementes |
580 |
( ¼ )1207 = 301.75 |
256,58 |
I s (P) |
|
Incolor |
Normal |
610 |
( ¼ )1207 = 301.75 |
314,89 |
i S (P) |
|
Incolor |
Com sementes |
8 |
( ¼ )1207 = 301.75 |
285,96 |
i s (R) |
|
Total |
|
1207 |
|
1141,45 |
|
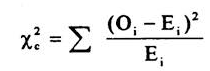 Logo X2 = 1141,45 GL = 4 -1 = 3.
Logo X2 = 1141,45 GL = 4 -1 = 3.
Consultando uma tabela
apropriada constatamos que a <
0,01%. Logo é pequeno o erro ao rejeitar a hipótese e independência e
concluímos que há evidências de ligação fatorial. O genótipo de P2 pode ser
estabelecido a partir dos gametas paternais (mais frequentes). Assim,
concluímos que: P2 = I
s // i S
b) Qual é
a distância entre os dois genes?
Análise da planta 1 – P1
|
Pericarpo |
Pendão |
Observado |
Tipo de gameta da P1 |
|
Vermelho |
Normal |
1219 |
I S (P) |
|
Vermelho |
Com sementes |
15 |
I s (R) |
|
Incolor |
Normal |
14 |
i S (R) |
|
Incolor |
Com sementes |
1174 |
i s (P) |
|
Total |
|
2422 |
|
d(I/i-S/s)= [100(R + R)]/Total = [100(15+14)]/2422 = 1,19 cM
Análise da planta 2 – P2
|
Pericarpo |
Pendão |
Observado |
Tipo de gameta da P2 |
|
Vermelho |
Normal |
9 |
I S (R) |
|
Vermelho |
Com sementes |
580 |
I s (P) |
|
Incolor |
Normal |
610 |
i S (P) |
|
Incolor |
Com sementes |
8 |
i s (R) |
|
Total |
|
1207 |
|
d(I/i-S/s)= )=
[100(R + R)]/Total = [100(8+9)/1207] = 1,4 cM
c) Qual é
a frequência esperada de plantas com pericarpo incolor e semente no pendão, na
descendência resultante:
c1) do
cruzamento entre as plantas 1 e 2?
Como
temos duas estimativas de distância entre os genes I/i e S/s, vamos admitir que
o valor mais provável da distância seja representado
pela média destas estimativas. Ou seja:
d =
(1,19 + 1,4)/2 =
1,3cM
Cruzamento:
|
P1 = I
S // i s |
x |
P2 = I
s // i S |
|
|
|
|
|
|
P(incolor, com semente) = P(ii ss) = ? |
|
Resposta:
P(iiss)
= P(is
de P1) x P(is de P2) = PxR
Sendo
P = (1-d)/2
= (1 – 0,013) /2 = 0,4935
R = d/2 = 0,013/2 = 0,0065
Assim
P(iiss)
= P(is
de P1) x P(is de P2) = 0,4935x0,0065= 0,00320775 = 0,32%
c2) da
autofecundação da planta 1?
Autofecundação:
|
P1 = I S // i s |
|
|
|
P(incolor, com semente) = P(ii ss) = ? |
Resposta:
P(iiss)
= P(is
de P1) x P(is de P1) = PxP
Sendo
P = (1-d)/2
= (1 – 0,013) /2 = 0,4935
R = d/2 = 0,013/2 = 0,0065
Assim
P(iiss)
= PxP
= 0,4935x0,4935= 0,24354 = 24,35 %
c3) da
autofecundação da planta 2? (4,225X10-5 ou 0,004225%)
Autofecundação:
|
P2 = I s // i S |
|
|
|
P(incolor, com semente) = P(ii ss) = ? |
Resposta:
P(iiss)
= P(is
de P2) x P(is de P2) = RxR
Sendo
P = (1-d)/2
= (1 – 0,013) /2 = 0,4935
R = d/2 = 0,013/2 = 0,0065
Assim
P(iiss)
= RxR
0,0065x0,0065 = 0,00004225 = 0,004225%
26) Um homem produz 5%
de espermatozoides AB. Pede-se:
a) Os genes A/a e B/b estão ligados, explique.
Sim. Esta baixa frequência é indicativo de que se trata de gametas
recombinantes.
- Quando não há ligação esperamos que todos os gametas (em
relação a dois genes) sejam produzidos
em igual frequência, ou seja: 25% AB, 25% Ab, 25% aB e 25% ab. Este fenômeno não foi verificado.
- Quando há ligação fatorial esperamos que sejam formados alguns
tipos de gametas em maior frequência (gametas paternais) e outros em menor
frequência (gametas recombinantes)
b) Qual é o genótipo desse homem?
Pela baixa frequência (5%) devemos admitir que o gameta AB é recombinante e, também, o gameta ab. Por outro lado, os gametas mais frequentes (paternais) deverão ser Ab e aB.
Desta forma, é coerente afirmar
que o genótipo do indivíduo possa ser reconstituído pelos seus gametas
paternais e, portanto, dado por: Ab//aB.
c) Qual é a frequência de gametas Ab produzidos por esse homem?
Sendo Ab um gameta
do tipo paternal sua frequência será igual a P. Devemos lembrar que:
2P + 2R = 100%
Como R =5%
Então
P = (100-10)/2
= 45%
d) Qual é a distância entre os genes A/a e B/b?
Sabemos da seguinte expressão : distância = % Recombinação
% recombinação=distância = [100(R + R)]/Total
Para fins de predição das
frequências de todos os gametas, fazemos Total = 100%. Assim,
d = [100(5 + 5)]/100 = 10 % = 10 cM
e) Durante a gametogênese, em que porcentagem
de células ocorre permuta entre os genes A/a e B/b?
A quantidade de células com
permuta poderá ser medida pela frequência de quiasmas. Quiasmas são pontos de
intercâmbios genéticos, considerados evidências citológicas da permuta. Assim,
%Quiasma (A/a – B/) = 2 [%Recombinação
(A/a – B/b)] = 2(10%) = 20%
27) Numa determinada
espécie, os genes A/a e B/b são ligados (10 cM de
distância). O cruzamento de um duplo-heterozigoto em fase de aproximação com um
duplo-recessivo produz descendentes AaBb
em que proporção?
Faremos primeiro o cruzamento de um duplo-heterozigoto em aproximação com um duplo recessivo, e realizar o cálculo das distâncias:
Cruzamento AB//ab x ab//ab
|
Gametas |
ab (100%) |
Frequencia |
|
AB P |
AB // ab |
(1-d)/2 = (1-0,1)/2=0,45 |
|
ab
P |
ab // ab |
(1-d)/2 = (1-0,1)/2=0,45 |
|
Ab R |
Ab // ab |
d/2 =
0,1/2 = 0,05 |
|
aB
R |
aB // ab |
d/2 =
0,1/2 = 0,05 |
Assim, constata-se que:
P(AaBb)=0,45x1=0,45
28) Os dois genes A/a e
D/d estão tão fortemente ligados que nunca se observou um recombinante. Se AAdd for cruzado com aaDD, qual é a proporção genotípica esperada na F2?
Primeiro realizamos o cruzamento dos indivíduos:
![]() Cruzamento: AAdd x aaDD
Cruzamento: AAdd x aaDD
F1: Ad // aD
(repulsão)
A população F2 será formada a
partir do encontro dos gametas representados a seguir:
|
|
Ad ( ½ ) |
aD ( ½ ) |
|
Ad ( ½ ) |
Ad // Ad |
Ad // aD |
|
aD ( ½ ) |
aD // Ad |
aD // aD |
Por estar completamente ligados, não temos recombinantes.
Assim a proporção genotípica na F2 será:
1 Ad //Ad :
2Ad //aD : 1aD//aD
29) Uma fêmea animal
com o genótipo AaBb é cruzada com um macho
duplo-recessivo. Sua prole inclui 442 AaBb,
458 aabb, 46 Aabb e 54 aaBb. Qual é a distância entre os dois genes
e qual é a sua fase de ligação na fêmea?
Inicialmente avaliaremos
se os genes são independentes:
Cruzamento: AaBb x aabb
|
Classes |
Gameta do duplo-heterozigoto |
Observado |
Esperado (Ho: genes A/a e B/b independentes) |
|
A a B b |
A B |
442 |
(1/4) 1000 = 250 |
|
a a b b |
a b |
458 |
(1/4) 1000 = 250 |
|
A a b b |
A b |
46 |
(1/4) 1000 = 250 |
|
a a B b |
A B |
54 |
(1/4) 1000 = 250 |
|
Total |
|
1000 |
|
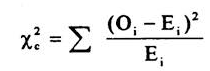
Logo
X2 = 640,64
GL = 4 -1 = 3.
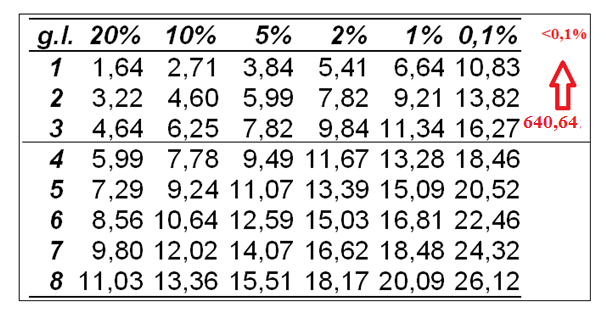
Conclusão: O valor de a < 0,001 indicado baixo erro ao rejeitar Ho.
Portanto, há evidências de que os genes estão ligados.
A distância entre os genes é
dada por:
d =[100(R + R)]/Total = [100(46+54)]/1000 =10cM
Portanto a fase de ligação na fêmea é de aproximação.